mikroRNA




Et mikroRNA (forkortet miRNA) er et lille ikke-kodende RNA-molekyle indeholdende ca. 22 nukleotider, der fungerer i den post-transkriptionelle regulering af genekspressionen, se proteinbiosyntesen. Det er små molekyler med stor effekt.[1] Det er foreslået at miRNA'er er dybt forbundet med og nødvendige for fremkomsten af komplekse hjerner.[2]
miRNA'er fungerer på mRNA-niveau primært via baseparring med komplementære sekvenser i mRNA-molekylerne. Som resultat bliver disse mRNA molekyler bragt til tavshed, hæmmet, på engelsk generelt kaldt “RNA silencing”. Dette sker for miRNA ved en eller flere af følgende processer:
(1) Spaltning af mRNA-strengen i to stykker
(2) Destabilisering af mRNA'et ved afkortning af dets polyA-hale
(3) Mindre effektiv translation på ribosom-niveau.
miRNA'er ligner andre små RNA’er som f.eks. de små interfererende RNA'er (siRNA'er) bortset fra at de foldes tilbage på sig selv for at danne korte hårnåle, mens siRNA'er stammer fra længere regioner med dobbeltstrenget RNA.
Det humane genom koder for mange miRNA'er, og der er med sikkerhed identificeret omkring 2600 gener for miRNA i mennesket og 162 i blæksprutter.[2][3]
Mange miRNA'er er evolutionsmæssigt bevaret, hvilket betyder, at de har vigtige biologiske funktioner. Dette kan betyde at miRNA indgår i reguleringen af så mange som 60% af menneskers og dyrs gener.[4] Deregulering eller malfunktion af mikroRNA har vist sig at være forbundet til udviklingsafvigelser, fysiologiske og adfærdsafvigelser og cancer.[5][6]
Mekanisme
Et miRNA er komplementært til en del af et eller flere messenger RNA (mRNA) og regulerer derved genudtrykket. miRNA fra mennesker og dyr er sædvanligvis komplementære til en del af 3' UTR -regionen af mRNA, hvorimod miRNAs fra planter sædvanligvis er komplementære til en del af den kodende region af mRNA. Perfekt eller næsten perfekt baseparring vil fremme den enzymatiske spaltning af mRNA (se en:Argonaute), som er den primære mekanisme hos planter.
OncomiR
En oncomiR (oncomikroRNA) forårsager cancer ved at nedregulere gener på to måder: ved hæmning af translationen eller ved at destabilisere mRNA. Særligt udsatte er generne, der regulerer cellens kredsløb. I et cancervæv kan mængden af en oncomiR være hævet eller sænket. Ved forhøjet niveau undertrykker oncomiR sandsynligvis et tumor suppressor-gen, se en:tumor suppressor. Ved sænket niveau sættes reguleringen fri og cellerne kan proliferere.
miRNA er også fundet hos virus som f.eks. hos Epstein–Barr virus, EBV, der er associeret til flere typer cancer.
Nogle eksempler
- miR-27, cancerregulering, se en:miR-27
- miR-31, metastaseregulering[7]
- miR-33, forhøjet lipoprotein, se en:miR-33
- miRNA-96, betydning af mutationer i miRNA[8]
- miR-122, oncogenregulering, se ill. og en:miR-122
- miRNA-335(-5p), oncogenregulering[9]
- miR-338, se en:miR-338
- miR-483-5p, stress-regulering[10]
Andre RNA
Oversigt over små, mindre og større RNA:
- anti-miR eller blockmir, se en:Antagomir
- ceRNA, se en:Competing endogenous RNA (CeRNA)
- circRNA (circulært RNA)
- crRNA, CRISPR RNA
- exRNA (extracellulært RNA)
- gRNA, guide-RNA
- lncRNA, se en:long non-coding RNA
- miRNA, mikroRNA eller miR
- piRNA se en:piwi-interacting RNA
- siRNA se en:small interfering RNA
- snRNA se en:small nuclear RNA
- snoRNA se en:small nucleolar RNA
- SNP RNA, signal recognition particle RNA
- tnRNA se en:tiny noncoding RNA
- tracrRNA, se en:Trans-activating crRNA
Se også
Eksterne links og henvisninger
- ^ MikroRNA - små gener med stort potentiale. Videnskab.dk 2009
- ^ a b Octopus Brains Evolved to Share a Surprising Trait in With Our Brains. ScienceAlert 2022
- ^ MicroRNA Target Recognition and Regulatory Functions. Cell. 2009
- ^ A Uniform System For The Annotation Of Human microRNA Genes And The Evolution Of The Human microRNAome. Annual Review of Genetics 2015
- ^ Biological principles of microRNA-mediated regulation: shared themes amid diversity. Nat. Rev. Genet. 2008
- ^ Advanced extraction of urinary microRNA for early cancer detection. Nature Portfolio 2021
- ^ RNA Snippet Suppresses Spread Of Aggressive Breast Cancer. Science Daily 2009
- ^ Targeted genome editing restores auditory function in adult mice with progressive hearing loss caused by a human microRNA mutation. Science 2024
- ^ MiRNA-335 suppresses neuroblastoma cell invasiveness by direct targeting of multiple genes from the non-canonical TGF-β signalling pathway. Carcinogenesis 2012
- ^ miR-483-5p offsets functional and behavioural effects of stress in male mice through synapse-targeted repression of Pgap2 in the basolateral amygdala. Nature 2024
Medier brugt på denne side
Forfatter/Opretter: A. Zinovyev, N. Morozova, A. N. Gorban, A. Harel-Belan, Licens: CC BY 3.0
Interaction of microRNA with protein translation process. Several (from nine documented) mechanisms of translation repression are shown: M1) on the initiation process, preventing assembling of the initiation complex or recruiting the 40S ribosomal subunit; M2) on the ribosome assembly; M3) on the translation process; M7, M8) on the degradation of mRNA. There exist other mechanisms of microRNA action on protein translation (transcriptional, transport to P-bodies, ribosome drop-off, co-translational protein degradation and others) that are not visualized here. Here, 40S and 60S are light and heavy components of the ribosome, 80S is the assembled ribosome bound to mRNA, eIF4F is an translation initiation factor, PABC1 is the Poly-A binding protein, and "cap" is the mRNA cap structure needed for mRNA circularization (which can be the normal m7G-cap or artificial modified A-cap). The initiation of mRNA can proceed in a cap-independent manner, through recruiting 40S to IRES (Internal Ribosome Entry Site) located in 5’UTR region. The actual work of RNA silencing is performed by RISC (RNA-induced silencing complex) in which the main catalytic subunit is one of the Argonaute proteins (AGO), and miRNA serves as a template for recognizing specific mRNA sequences.
Forfatter/Opretter: VTD, Licens: CC BY-SA 4.0
Examples of miRNA stem-loops, with the mature miRNAs shown in red. Shown are predicted stem-loop secondary structures within the pri-RNA molecules that give rise to the lin-4 miRNA of C. elegans and the miR-1 miRNA of humans. This figure was drawn based on studies that cloned and sequenced mature miRNAs and predicted the corresponding stem-loops. [1][2]
Forfatter/Opretter: Opabinia regalis, Licens: CC BY-SA 3.0
The secondary structure of a precursor microRNA sequence from Brassica oleracea, as predicted by MFOLD (http://bioweb.pasteur.fr/seqanal/interfaces/mfold-simple.html) by M. Zuker. GIF generation by plt22gif, D. Stewart and M. Zuker 2007.
(c) Narayanese at engelsk Wikipedia, CC BY-SA 3.0
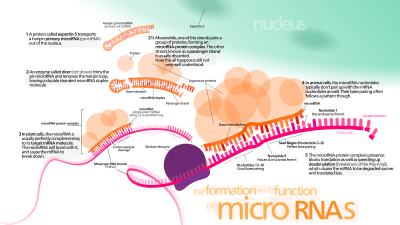
Overview of microRNA processing in animals, from transcription to the formation of the effector complex. There are two pathways, one for microRNAs from independent genes and one for intronic microRNAs.
Enzymes in the picture:
Drosha, Pasha (pri-miRNA → pre-miRNA)
Spliceosome (pre-mRNA → intron lariat)
Debranching enzyme (intron lariat → RNA that can fold into pre-miRNA)
RAN-GTP, Exportin-5 (export from nucleus)
Dicer (pre-miRNA → miRNA)
pri-miRNA = primary microRNA transcript
pre-mRNA = precursor messenger RNA
pre-miRNA = precursor microRNA
miRNA = microRNA
miRNA* = antisense microRNA
miRNP = microRNA ribonucleoprotein
miR-122 secondary structure and sequence conservation






