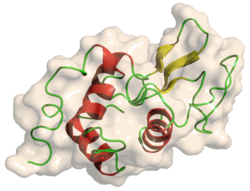
Lysozym



Substratet (sort)
Bindingssædet (blåt)
Det aktive sæde (rødt)

Lysozym er et enzym, der fungerer ved at hydrolysere og bryde glykosidbindinger i peptidoglykaner. Enzymet kan også bryde glykosidbindinger i chitin, men ikke så effektivt som specifikke chitinaser. Lysozym kaldes også muramidase eller N-acetylmuramid glycanhydrolase og er et antimikrobielt enzym, der findes hos mennesker og dyr, hvor det udgør en del af det medfødte immunsystem.
Lysozym findes rigeligt i sekreter som tårer, spyt, modermælk og slim. Det er også til stede i cytoplasmatiske granula af makrofager og polymorfonukleære neutrofiler (PMN'er).
Store mængder lysozym findes i æggehvide.
Lysozym var den anden proteinstruktur (efter myoglobin) og den første enzymstruktur der blev løst.[1]
Mekanisme
Lysozym er en glycosidhydrolase, der katalyserer hydrolysen af 1,4-beta-bindinger mellem N-acetylmuraminsyre (NAM) og det fjerde carbonatom i N-acetylglucosamin (NAG) i peptidoglycaner, som er hovedkomponenten i gram-positive bakteriers cellevæg. Bindingen af peptidoglycan-molekylet sker i en kløft mellem lysozyms to domæner.
Historisk
Den antibakterielle egenskab af æggehvide blev først observeret af Laschtschenko i 1909. Den bakteriedræbende aktivitet af næseslim blev demonstreret i 1922 af Alexander Fleming, opdageren af penicillin, som gav enzymet navnet lysozym: "Da dette stof har egenskaber, der ligner fermenternes egenskaber, har jeg kaldt det 'Lysozym'." Fleming fortsatte med at vise, at et lysozym var til stede i en lang række sekreter og var i stand til hurtigt at lysere forskellige bakterier, især en gul kok, som han arbejdede med.
Æggehvide-lysozym blev først krystalliseret af Edward Abraham i 1937, hvilket gjorde det muligt for David Chilton Phillips i 1965 ved hjælp af røntgenkrystallografi at udarbejde den tredimensionale struktur med en opløsning på 2 Ångström.
Lysozym var således den anden komplette proteinstruktur og den første enzymstruktur, der blev løst. På grundlag af den tredimensionale struktur var lysozom da også det første enzym, der fik beskrevet en detaljeret, specifik mekanisme, hvoraf der kunne udledes en generel forståelse af, hvordan enzymer fremskynder en kemisk reaktion ud fra atomers placering i den fysiske struktur.
Henvisninger
Medier brugt på denne side
Forfatter/Opretter: Thomas Shafee, Licens: CC BY 4.0
Organisation of enzyme structure and lysozyme example. Binding sites in blue, catalytic site in red and peptidoglycan substrate in black. (PDB: 9LYZ)
Forfatter/Opretter: No machine-readable author provided. Lode~commonswiki assumed (based on copyright claims)., Licens: CC BY-SA 3.0
Single Protein crystal of Lysozyme
Forfatter/Opretter: Yikrazuul, Licens: CC BY-SA 3.0
structure of Lysozyme, based on X-ray structure (Organism: Gallus gallus) pdb 132L
Forfatter/Opretter: Pierreb24, Licens: CC0
Hydrolysis β-1, 4-glycosaminidasic of petidoglycan, main constituant of some bacteria's cell menbrans. Cleavage is done specificly between NAG (n-acetylglucosamin) and NAM (N-actelymuramic acid).